新朋友点上方蓝字“蛋白质组代谢组学”快速关注
蓝藻去酰基化酶CddA研究历程回顾
刘欣
武汉轻工大学

岁月流转,时光如流水轻烟,转眼间我从葛老师实验室毕业已逾一年。看着旁边熟睡的女儿,回顾着读博期间的点点滴滴,突然感觉自己是世界上最幸运的人,感激身边的老师、朋友和家人对我的无私的照顾和帮助。在此分享我读博期间的一段经历,希望能够将这份幸运传递给身边的每一个人。
2014年夏天,为了不荒废大学暑假的时光,我来到了葛峰老师实验室实习,葛老师安排王炎师兄和陈卓师兄教我基本的实验技能。这是我第一次系统地学习做生物学实验,也是我第一次认识蛋白质翻译和修饰,也就是在这时与蛋白质翻译后修饰结下了不解之缘。后来,我跟葛老师申请考实验室的硕士研究生,葛老师人和蔼可亲,很欢迎我报考,最后我也顺利地考入了中科院水生所,开始了我幸运的求学生涯。
2016年6月, 我大学毕业后来到了葛老师实验室提前开始研究生的工作,从王炎师兄那里接手了蓝藻赖氨酸琥珀酰化修饰组学的研究工作。正式进入实验室的一个月,我跟着马炎炎师兄学习蛋白质组学相关的基础知识和琥珀酰化修饰组学数据的处理技能。马炎炎师兄为人风趣幽默,还很有耐心,哪怕一个问题问几次他也会不厌其烦地解答,特别感激他对我的帮助和指导。随着蓝藻赖氨酸琥珀酰化修饰组学工作的开展,我的研究生生活也走上了正轨。
1. 悬而未决,不知所措
我们实验室一直致力于蓝藻翻译后修饰组学的研究。在我来之前,实验室已经在蓝藻中发现了很多翻译后修饰,例如乙酰化、磷酸化等等,但是一直有一个问题始终没有得到解决——蓝藻中是否存在调控蛋白质翻译后修饰的酶?实验室的师兄们做了许多努力,例如预测到了16个可能的赖氨酸乙酰转移酶和5个可能的赖氨酸去乙酰化酶,但是怎么验证是个大问题。一开始他们尝试过表达和纯化可能的乙酰转移酶和去乙酰化酶,与底物反应,并检测反应前后底物的修饰水平变化。很快,他们发现这条路走不通,因为酶与底物具有特异性,在既无法确定酶也无法确定底物的条件下,想要酶和底物能够发生反应无异于大海捞针。后来,他们又构建了这些可能的乙酰转移酶和去乙酰化酶的蓝藻突变株,期望从突变株体内的修饰水平变化中找到答案,但是由于一些反应性乙酰衍生物,包括乙酰磷酸、乙酰辅酶A等,也可以通过非酶机制使蛋白发生乙酰化修饰,使得蓝藻体内具有很高的乙酰化修饰背景,导致突变株体内的修饰水平没有显著的差异(图1),也就无法明确哪一个才是调控乙酰化修饰的酶。因此,这个课题也就此搁置。
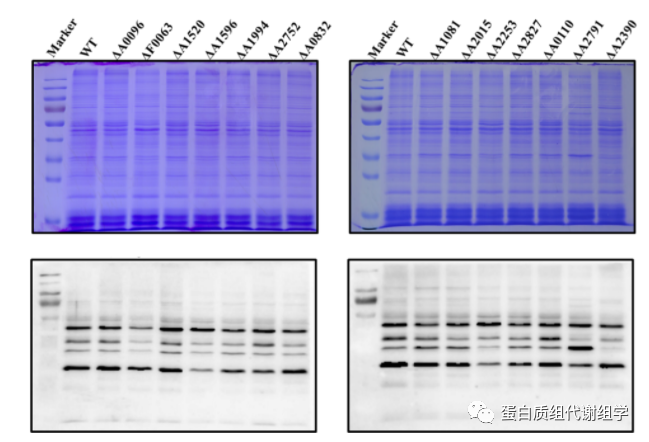
图1.Western blot验证可能的乙酰转移酶/去乙酰化酶敲除株乙酰化蛋白水平变化情况
2. 另辟蹊径,柳暗花明
在开展蓝藻赖氨酸琥珀酰化修饰组学的工作中,葛老师希望能够找到调控琥珀酰化修饰的酶,于是又回到了鉴定蓝藻中调控翻译后修饰酶的这个课题。在师兄们的试错中,我觉得之前的方法没有必要继续尝试了。在跟葛老师和杨明坤老师反复商量后,决定采用荧光底物的方法来筛选去酰基化酶,该方法主要是基于一个荧光基团AMC(7-amino-4-methylcoumarin),在非游离状态下,AMC只有很低的荧光,而在游离的状态下具有很强的荧光。我们设计合成带有不同修饰基团(Ac/Pr/Mal/Su/Glu)的荧光底物Boc-Lys(Acyl)-AMC;Boc为叔丁氧羰基保护基,用于保护氨基;Lys(Acyl)即为带有修饰基团的赖氨酸(图2A)。我们体外过表达并纯化预测的5个去酰基化酶(A2827,A0319,A1994,A2791,A1628)去与荧光底物反应,当Boc-Lys(Acyl)-AMC上赖氨酸的修饰基团被去酰基化酶催化去除后,胰酶(Trypsin)就能够切开Boc-Lys-AMC上Lys与AMC之间的酰胺键,AMC就会被释放出来从而发出很强的荧光。幸运的是实验结果发现A2791具有很强的去乙酰化酶的酶活和去丙酰化酶的酶活,我们也就将其命名为CddA(Cyanobacterial Deacetylase/Depropionylase)(图2B)。遗憾的是,这次实验并没有找到去琥珀酰化酶(图2C)。
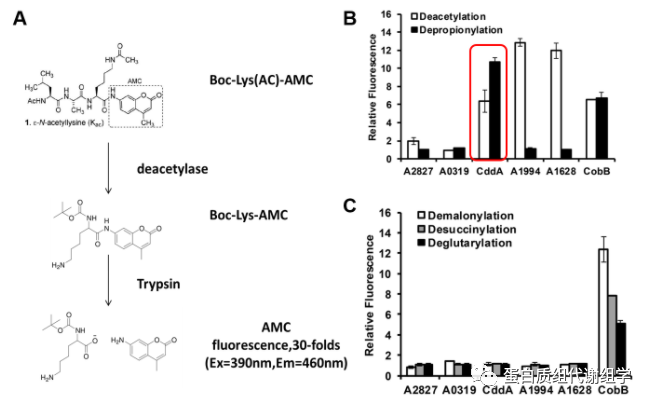
图2.荧光底物验证CddA具有去乙酰化酶和去丙酰化酶的酶
3. 抽丝剥茧,豁然开朗
初步确定了CddA的去酰基化酶功能后,为了进一步探索CddA的催化机制,我们决定对CddA进行晶体结构的解析。这是实验室之前从未做过的方向,对我而言是一个巨大的挑战。因此,葛老师帮忙联系了中山大学的刘迎芳教授协助我们开展这项工作。幸运的是,CddA是一个很容易纯化的蛋白,得到它的蛋白晶体并不算难,之后我们就将CddA的蛋白晶体送到了上海同步辐射光源进行X射线衍射数据的收集。数据回来之后,怎么分析也是令人头疼的问题。
在这里要特别感谢刘迎芳教授,给了我们很多数据分析上的宝贵建议,帮助我们解析了酶的活性中心。同时,中科院物理与数学研究所的李涛博士也给予我很大的帮助,他发给我很多pymol软件的学习资料,并教会我怎么去分析CddA的底物结合口袋,在此特别感谢各位老师的悉心教导和帮助。通过对CddA晶体结构的分析,我们发现CddA具有独特的α/βfold结构,并初步确定了CddA的催化活性中心(His126、His127、Phe136、Tyr192、Leu255和Tyr287)。通过点突变CddA催化活性中心位点,并检测其突变前后的CddA去酰基化水平,我们进一步验证了His126、His127、Phe136和Tyr287在调节CddA的催化活性中起重要作用。
虽然我们明确了CddA的催化调控机制,但是CddA在蓝藻内会发挥什么样的生理功能仍然是未知的。研究一个蛋白功能最经典的方法就是构建敲除株测定它的表型。因此,我们构建了CddA基因敲除株,并测定了CddA敲除株的一系列表型,包括蓝藻的生长曲线,电子传递速率等等。在这里特别感谢赵进东院士一直以来的指导,尤其是在测定电子传递速率的时候,赵院士在百忙之中能够抽出时间亲自指导我的实验,让我在实验过程中少走了许多弯路。
我们发现敲除CddA对聚球藻PCC 7002的生长和光合电子传递有显著的影响,而CddA又是一个去乙酰化和去丙酰化酶,为了进一步探究CddA是如何调控蓝藻的生理过程,我们针对聚球藻PCC 7002中的乙酰化蛋白、丙酰化蛋白以及CddA的作用靶标开展研究。考虑到之前我们实验室已经开展过蓝藻乙酰化组学的研究,为了避免工作上的重复,我们就在蓝藻中开展了赖氨酸丙酰化组学的工作。对于已经有过赖氨酸琥珀酰化组学研究经验的我而言,开展这部分工作十分顺利。我们利用高效的富集策略与高分辨质谱技术,共鉴定到了598个丙酰化修饰位点和382个丙酰化修饰蛋白,并通过生物信息学分析发现丙酰化修饰参与了聚球藻PCC 7002的碳代谢和光合作用。为了研究CddA在聚球藻PCC 7002中的调控机制,找到CddA的作用靶标是十分有必要的。怎么找到CddA的底物呢?首先是找能够发生乙酰化和丙酰化修饰的蛋白,也就是从乙酰化组学和丙酰化组学的数据中筛选,但是一共有896个蛋白可以发生酰基化修饰,一个个验证效率太低了,怎么进一步缩小范围呢?通过与葛老师、杨老师讨论,我们一致认为CddA的底物修饰水平在ΔCddA敲除株中肯定是要比野生型中高。因此我们就从这一点出发去缩小底物的范围,也就是用赖氨酸乙酰化/丙酰化修饰抗体对聚球藻PCC 7002野生型和ΔCddA敲除株中的总蛋白乙酰化/丙酰化修饰水平进行了Western blot分析进而寻找修饰差异的蛋白,结果我们在40-55kDa之间找到了一个蛋白条带,其乙酰化和丙酰化修饰水平变化显著,所以我们猜测在该位置处可能存在CddA的作用靶标。我们就对这一位置的蛋白条带进行了胶内酶解和质谱鉴定,找到了很多功能重要的蛋白质。我们将这些蛋白与修饰组学鉴定到的修饰蛋白取交集,就发现了一个功能重要的蛋白质F/SBPase。F/SBPase具有果糖-1,6-二磷酸酶和景天庚酮糖-1,7-二磷酸酶的酶活,是糖酵解中的限速酶。我们推测CddA可以调控F/SBPase进而调控其酶活。为了验证这一猜想,我们就对F/SBPase与CddA反应前后的乙酰化/丙酰化修饰水平和酶活水平进行了检测。结果正如我们猜想的一样,CddA能够催化糖代谢中的关键酶果糖-1,6-二磷酸酶(F/SBPase)的赖氨酸去乙酰化和去丙酰化过程,从而实现对其酶活性的调控作用。
4. 百尺竿头,再进一步
在2018年10月,我基本完成了论文的实验部分,开始参与到论文的写作中,但写作过程没有想象的顺利。一是我的写作水平较差,这是导致效率较低的主要原因。经常把写好的部分发给杨老师,杨老师又返回来要求重写,一小部分内容经常反复修改。二是写CddA晶体结构这部分结果的时候,由于我对晶体结构这一领域的研究不太熟悉,写起来觉得十分困难,进度非常缓慢,经常一天只憋出来一句话。但是在deadline的鞭策下,只能每天在实验室看这方面的文献,梳理这一部分结果的框架。虽然这些功课加深了我对这部分结果的理解,但是动起笔来还是不尽人意。还好这部分内容最后得到了刘迎芳教授的详细指导和协助修改,从中我也收获颇丰。
2019年2月19日,论文终于提交到PNAS编辑部。经过一个月的漫长等待,我们在2019年3月20日却收到了PNAS的拒稿信。拒稿的主要原因是第一个审稿人认为,赖氨酸乙酰化主要是由于乙酰磷酸的积累引起的,乙酰化不针对特定的代谢途径,而只是针对可获得的赖氨酸,因此他不认为广泛的赖氨酸酰基化修饰是调控机制的一部分,想要证明翻译后修饰对蓝藻光合作用和碳代谢的全局调控至少还需要两个证据:一是根据环境变化加上或去除翻译后修饰的生理条件,二是发现给赖氨酸加上乙酰基或丙酰基基团的酶。审稿意见回来之后,我陷入了失望和迷茫。毕竟我们实验室之前在寻找酰基转移酶上已经做了许多努力,但是还是没有结果,因此我觉得我能找到酰基转移酶的可能性也不大,这就回复不了第一个审稿人的问题。跟葛老师商量之后,葛老师认为编辑和第二个审稿人的意见很正面,还是想再试着找一下酰基转移酶,设法回答第一个审稿人的问题。葛老师的话又让我燃起了斗志。于是,我们开始检索鉴定酰基转移酶方法的相关文献,结果我们惊喜地发现芝加哥大学的Wolfe教授刚刚在mBio上发表了一篇文章,这篇文章创建了一个大肠杆菌乙酰转移酶筛选系统,该系统主要是由一个极低乙酰化修饰背景的突变体(Negative Control,NC)搭建而成的。
根据细胞内乙酰化的调控机制,该突变体缺失了已知的细菌乙酰转移酶PatZ、非酶途径反应性乙酰衍生物AcP生成途径的关键酶磷酸乙酰转移酶(Phosphate acetyltransferase,Pta),以及去乙酰化酶CobB,使其在保持极低乙酰化修饰背景的同时也保持了细胞内乙酰化修饰水平的相对稳定。Wolfe教授通过在该突变体(NC)中过表达大肠杆菌中可能的乙酰转移酶,并检测表达前后乙酰化修饰水平的变化,来验证其是否具有乙酰转移酶的酶活,最终发现了5个大肠杆菌乙酰转移酶。认真研读了这篇文章后,我马上去找葛老师讨论,我们一致认为把Wolfe教授的这个系统用来筛选蓝藻中的乙酰转移酶是可行的。因此,葛老师立马安排我与Wolfe教授取得联系。幸运的是,Wolfe教授很快给了我回信,并愿意将乙酰转移酶筛选系统相关的大肠杆菌突变体邮寄给我们。突变体一到,我立马用这个系统来筛选蓝藻中的乙酰转移酶,看最终WB结果的时候,我激动地发现,竟然真的找到了蓝藻中乙酰转移酶(图3)!我立马把这个好消息分享给了杨老师和葛老师。找到了乙酰转移酶,我们认为文章回复回去,PNAS接收的可能性很大,但令我们大失所望的是,第一个审稿人仍然拒稿了。
我们只得整理心情转投Plant Physiology,2020年1月19号Plant Physiology审稿意见回来了,审稿意见还比较乐观,但是需要补一个CddA点突变与F/SBPase反应前后的乙酰化WB。这时恰逢武汉疫情最严重的时候,我被封控在老家回不去武汉,无法进行补充实验,只能在家干着急。这时杨老师挺身而出,不顾危险从襄阳一路开车回到武汉,将这一实验补充完整。最终文章在2020年5月8号修回,于2020年7月8号正式录用。
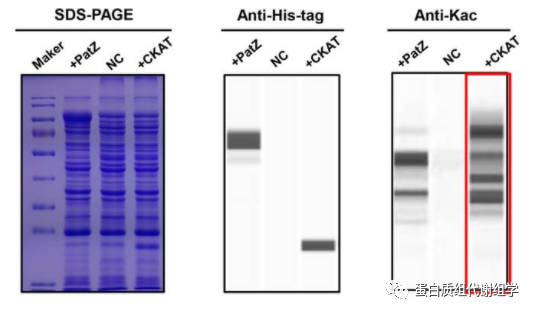
图3. Wes全自动蛋白质印迹定量分析系统(ProteinSimple)检测CKAT在低乙酰化修饰背景的突变体(NC)中过表达前后乙酰化修饰水平的变化。
5. 不忘初心,砥砺前行
时光荏苒,我已从当初青春年华的学生成长为一名高校教师。回首这段难忘的人生旅程,万千思绪涌上心头,我无比庆幸在成长的关键时刻遇到了葛老师,并在葛老师的引领下笃学笃行、不断精进,在蓝藻翻译后修饰研究中取得了突破性成果,让我不仅收获了一篇高质量的文章,对蛋白质翻译后修饰的调控机制有了更深的认识,还找到了蓝藻中的首个乙酰转移酶。更幸运的是,今年我也凭借乙酰转移酶这个课题申请到了青年基金。毫无疑问,葛老师的悉心培养对我而言异常珍贵,在科学实验的态度、研究学习的思路、深入科研的能力等各个方面的成长和提升无不是在葛老师的教导下才得以实现。
除此之外,葛老师还在人生成长的更深处影响着我。他的科研成就我不能望其项背,但那严谨治学、明德惟馨的高尚品行一直深深牵引着我的学习成长、科学研究和教学工作,滋养着我人生发展的每一步。
如今,从葛老师实验室光荣毕业后的我仍然从事着蛋白质组学的研究工作。我无比热爱该领域的研究工作,我将继续扎根深入在里面,站在葛老师的巨人肩膀上寻求新的突破;我将以葛老师为灯塔,带领有志于此的青年学生们在生命科学的浩瀚大海中乘风破浪,开辟出属于我们的更精彩的新天地。

2022年12月5日写于东湖之滨
liuxin4550@126.com
鸣谢
感谢北京欧米科斯生物科技有限公司作为独家赞助商对本公众号的大力支持!

发表评论